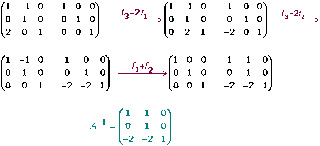He estado a punto de titular este artículo Pretty emacs, como el artículo de Alexandre Vassalotti. Creo que es muy acertado, pero he preferido no copiarlo.
He estado a punto de titular este artículo Pretty emacs, como el artículo de Alexandre Vassalotti. Creo que es muy acertado, pero he preferido no copiarlo.Emacs-snapshot es un conjunto de paquetes de la versión CVS de GNU Emacs, el conocido editor (y otras cosas), especialmente concebidos para la distribución de linux Debian y, por extensión, para Ubuntu. Han preparado dos versiones. Una para el sistema gráfico GTK y la otra para la consola. Yo utilizo la primera.
A poco que se navegue un poco por la red, todo el mundo coincide que Emacs es un potentísimo editor (dicen que incluso una forma de vida), pero también hay coincidencia en la dificultad de su aprendizaje. Así, muy pocos serán los que se atreban a probarlo. En Kubuntu hay una alternativa que es RKWard, mucho más sencillo i intuitivo, pero también ligeramente inestable. De modo que yo me animé a probar Emacs-snapshot.
Para trabajar con R, la instalación de Emacs-snapshot es muy sencilla:
sudo apt-get install emacs-snapshot ess
El segundo paquete es Emacs speaks statistics, imprescindible para que Emacs y R se entiendan (se supone que R está instalado).
Con esta instrucción se instalaran las versiones de los repositorios oficiales de Ubuntu. Si alguien desea lo último de lo último, hay que instalar los repositorios PPA for Ubuntu Emacs Lisp.
Una vez instalados, cuando abrimos un archivo con extensión .R, Emacs-snapshot lo reconoce como tal y la primera vez que utilicemos los botones de ejecución, abrirá una ventana para solicitar la carpeta de trabajo. La fijamos y aparecerá un buffer (ventana en el lenguaje de Emacs) con R en la parte inferior (ver la figura de arriba). En esta situación ya podemos ir ejecutando las instrucciones del archivo y viendo los resultados en el buffer inferior. El sistema es sencillo y muy práctico. Y a partir de aquí, buscar un manual de Emacs o seguir su tutorial. El esfuerzo vale la pena.
Por cierto, ¿a qué viene lo de pretty emacs?
Pues, resulta que es muy fácil cambiar la fuente y poner la que más nos guste. La que viene por defecto suele ser horrorosa. Ver el post de Alexandre Vassalotti.
¿Y los que trabajamos con Windows? Pues a la izquierda tenéis un enlace a la, a mi parecer, más sencilla instalación para Windows.